Contact
COORDONNATEUR SCIENTIFIQUE
Pr Bernard LA SCOLA
bernard.la-scola@univ-amu.fr
RESPONSABLE TECHNIQUE
Philippe DECLOQUEMENT
philippe.decloquement@ap-hm.fr
Composition de l’équipe
L’équipe biobanque est composée de :
- 1 PU-PH
- 1 PU
- 4 ingénieurs
PRÉSENTATION
La plateforme d’analyses séparatives, appelée plus communément plateforme protéomique, de l’IHU est spécialisée dans l’analyse moléculaire hors acides nucléiques, de tous les agents pathogènes viraux, bactériens et parasitaires ainsi que leur cellules cibles, leur compositions, les composés qu’ils produisent.
Ce laboratoire est coordonné pour le compte de la fondation par le Pr Bernard La Scola sur le plan organisationnel et par un ingénieur AP-HM Philippe Decloquement sur le plan technique. La plateforme se partage entre les personnels de l’Université et de l’AP-HM, notamment pour ce qui relève du diagnostic. Il occupe tout l’aile ouest du 2ème étage du bâtiment des laboratoires de l’IHU Méditerranée infection.
Cette plateforme se caractérise par un imposant plateau technique autorisant schématiquement d’analyser sur un plan moléculaire toutes les compositions en glucides, lipides et protéines de toutes les biomolécules, notamment déterminer leur structure et composition moléculaire.
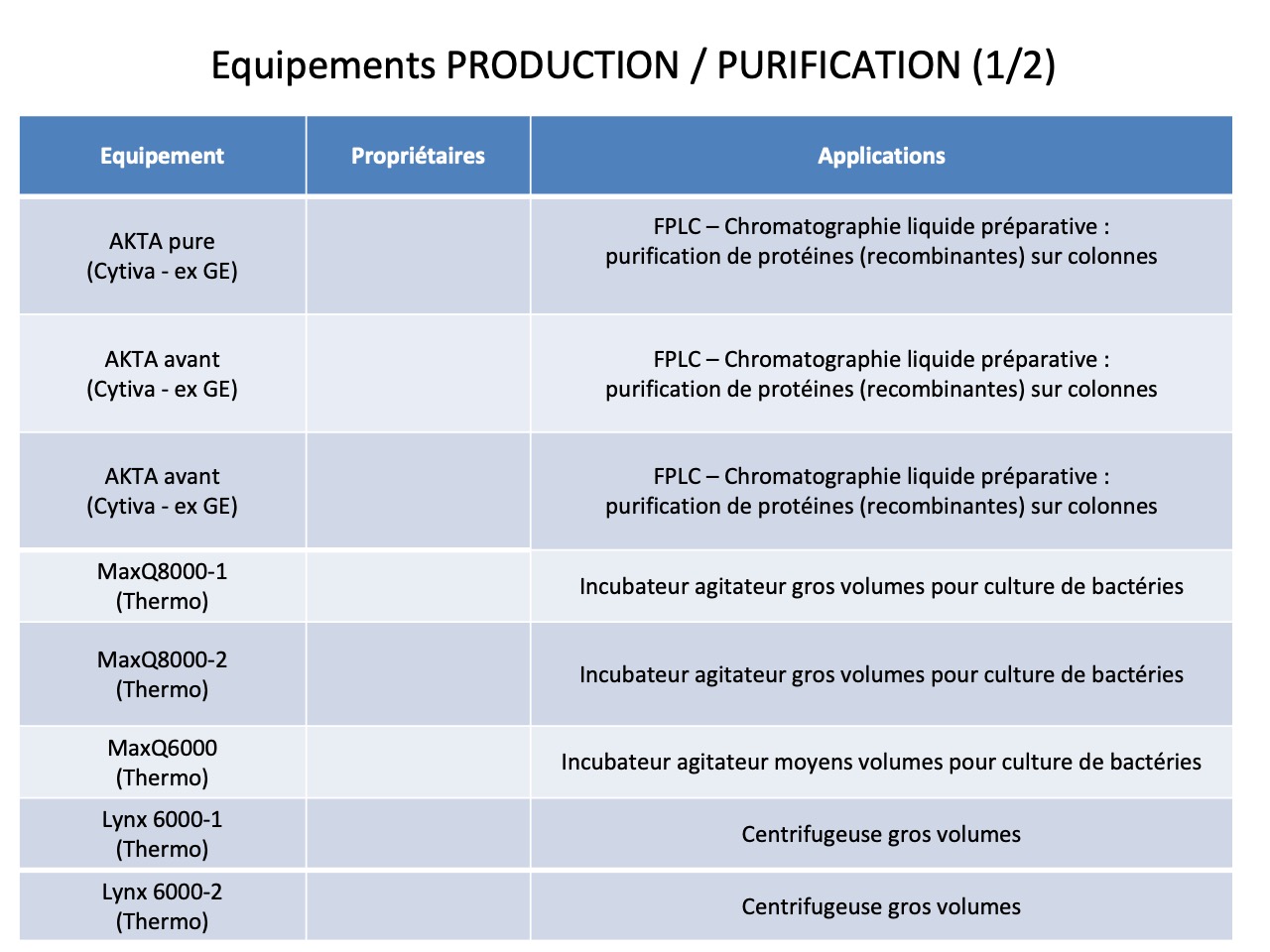
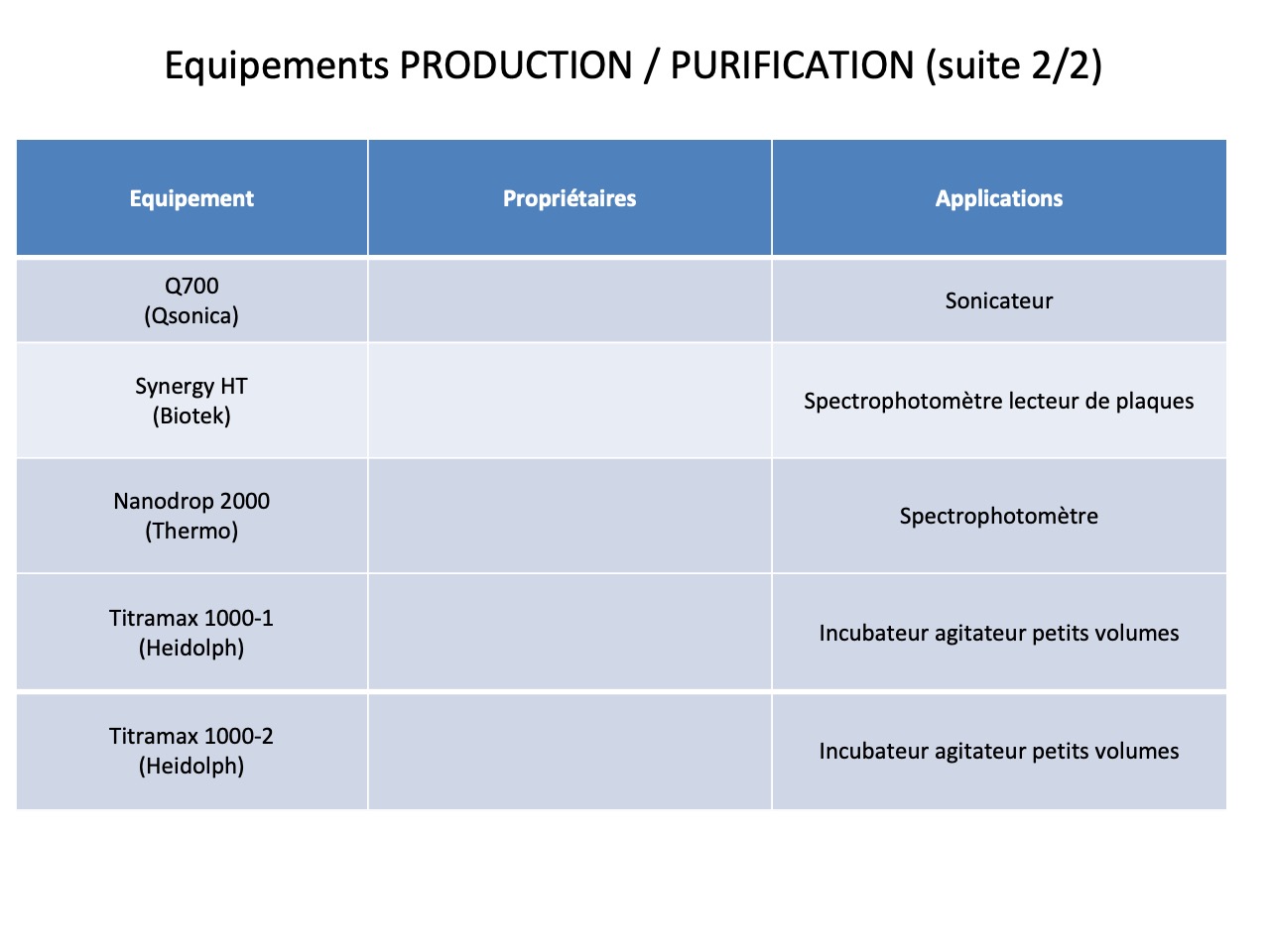
La plateforme peut aussi assurer les clonage de gènes puis l’expression in vitro de protéines à plus ou moins grange échelle, avec possibilité de productions à hauts débits à l’aide de fermenteurs.
Manipulant beaucoup de produits chimiques, elles est équipé de matériel permettant d’assurer la sécurité des personnels.
EXPERTISE
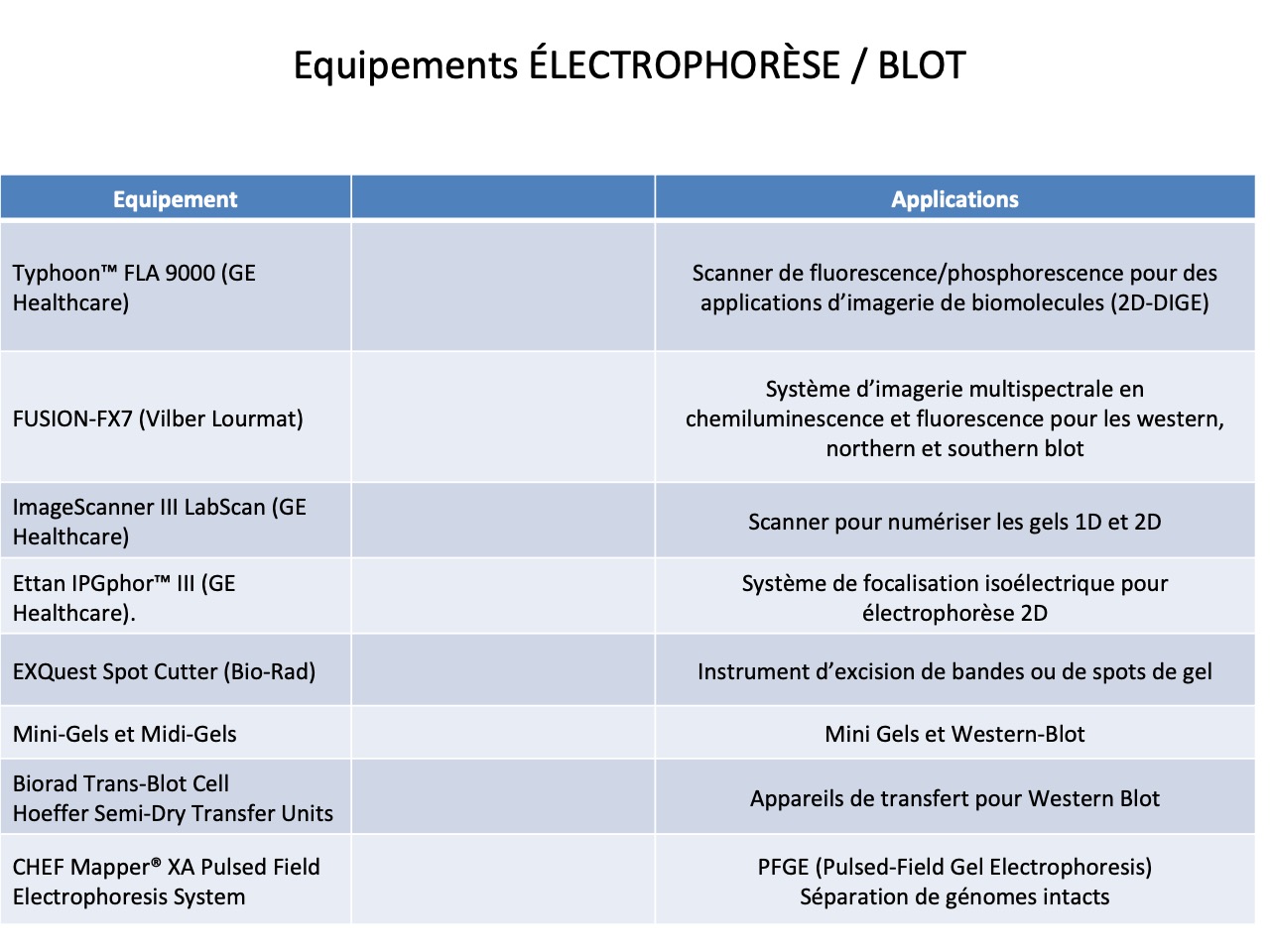
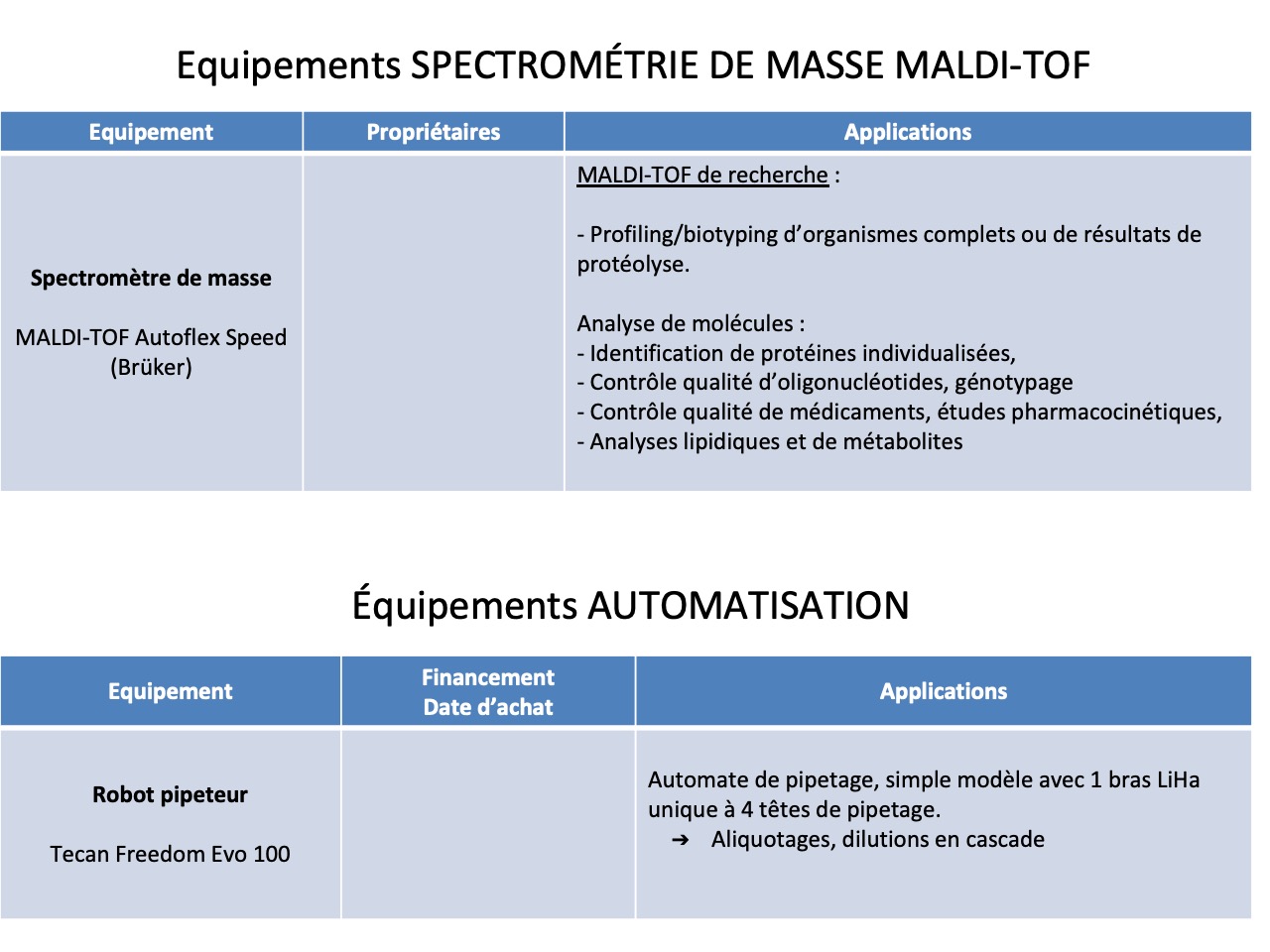
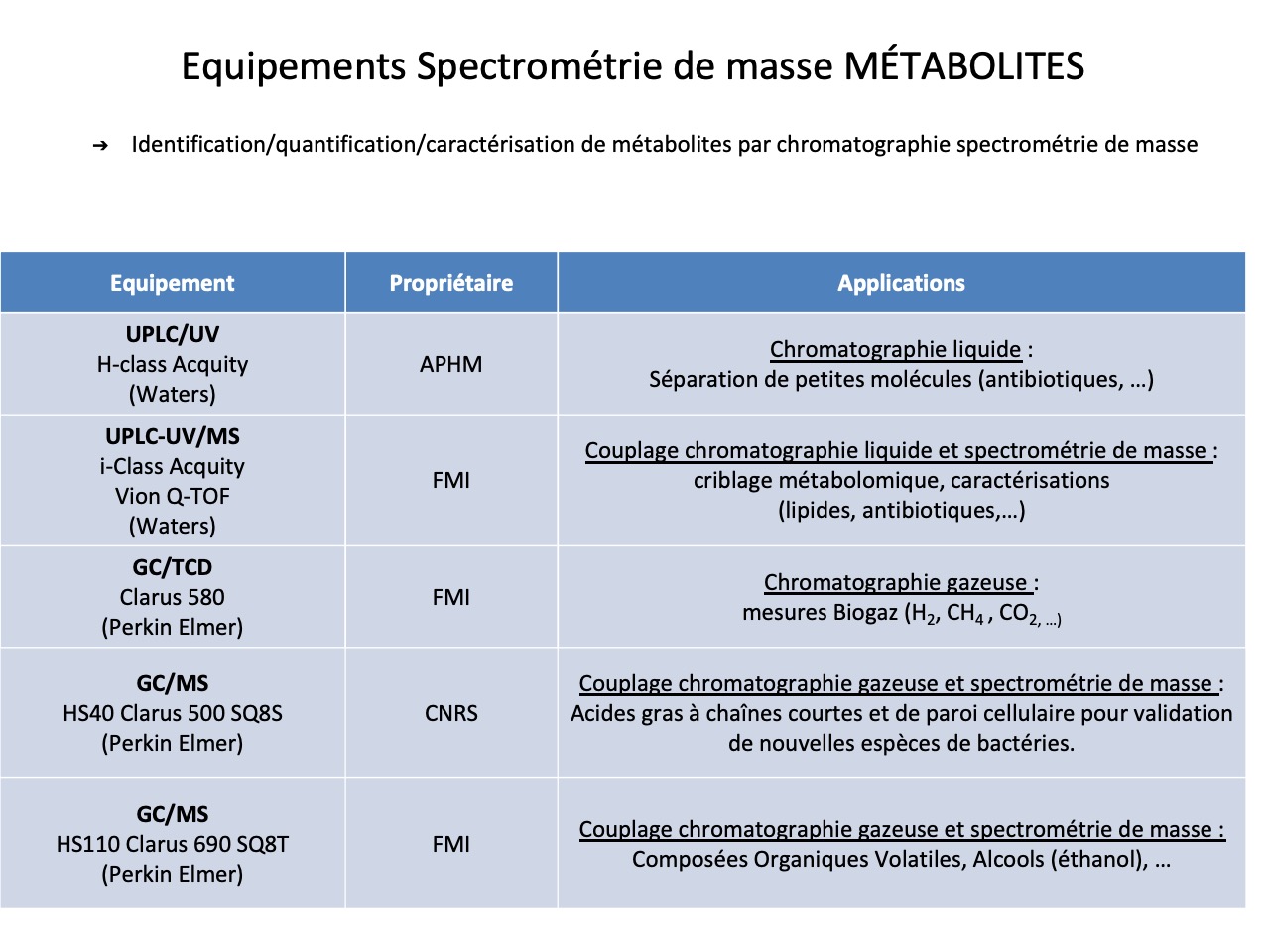
Le laboratoire possède les expertises en lien avec ses équipements. Equipement d’électrophorèse et blot (Western, Southern and Northern), electrophorèse 2-D ou champs pulsé. Equipements de purification et production, notamment biofermenteurs, incubateurs-agitateurs, centrifugeuses et ultracentrifugeuses gros volumes, 3 appareils de chromatographie liquide préparative, 2 spectrophotomètres lecteurs de plaques. Spectromètre de masse maldi-tof haute définition et plusieurs spectromètre de masse maldi-tof à haut débit pour identification de bactéries, levures et arthropodes. Des équipements de spectrométrie de masse pour analyse de métabolites, notamment chromatographie liquide couplée ou non à un appareil de spectrométrie de masse, chromatographie gazeuse couplée ou non à un appareil de spectrométrie de masse.
ÉQUIPEMENT ET TECHNOLOGIES
PRINCIPALES RÉALISATIONS
1: Charlier P, Augias A, Weil R, Bouchet F, Poupon J, Popescu MS, Decloquement P, Azza S, Angelakis E, Richardin P, Colson P, Dubourg G, Million M, Raoult D.
Scurvy complicated with Capnocytophaga sputigena sepsis as a possible cause of death of king Saint-Louis of France (1270 AD). Microb Pathog. 2023 Dec;185:106399. doi: 10.1016/j.micpath.2023.106399. Epub 2023 Oct 24. PMID:37884212.
2: Aherfi S, Brahim Belhaouari D, Pinault L, Baudoin JP, Decloquement P, Abrahao J, Colson P, Levasseur A, Lamb DC, Chabriere E, Raoult D, La Scola B. Incomplete tricarboxylic acid cycle and proton gradient in Pandoravirus massiliensis: is it still a virus? ISME J. 2022 Mar;16(3):695-704. doi: 10.1038/s41396-021-01117-3. Epub 2021 Sep 23. PMID: 34556816; PMCID: PMC8857278.
3: Sahmi-Bounsiar D, Baudoin JP, Hannat S, Decloquement P, Chabrieres E, Aherfi S, La Scola B. Generation of Infectious Mimivirus Virions Through Inoculation of Viral DNA Within <i>Acanthamoeba castellanii</i> Shows Involvement of Five Proteins, Essentially Uncharacterized. Front Microbiol. 2021 Jul 9;12:677847. doi: 10.3389/fmicb.2021.677847. PMID: 34305841; PMCID: PMC8299487.
4: Kieu HT, Garrigou N, Fadlane A, Brechard L, Armstrong N, Decloquement P, Yasir M, Azhar EI, Al-Masaudi SB, Lagier JC, Tidjani Alou M, Raoult D. Clostridium culturomicium sp. nov. and Clostridium jeddahitimonense sp. nov., novel members of the Clostridium genus isolated from the stool of an obese Saudi
Arabian. Curr Microbiol. 2021 Sep;78(9):3586-3595. doi: 10.1007/s00284-021-02616-4. Epub 2021 Jul 23. PMID: 34297170.
5: Ayona D, Zarza SM, Landemarre L, Roubinet B, Decloquement P, Raoult D, Fournier PE, Desnues B. Human galectin-1 and galectin-3 promote <i>Tropheryma whipplei</i> infection. Gut Microbes. 2021 Jan-Dec;13(1):1-15. doi: 10.1080/19490976.2021.1884515. PMID: 33573443; PMCID: PMC7889132.
6: Hasni I, Armstrong N, Decloquement P, Azza S, Fontanini A, Abbe O, Cherif Louazani A, Demanèche S, Chabrière E, Colson P, La Scola B. Proteomics and
Lipidomics Investigations to Decipher the Behavior of <i>Willaertia magna</i> C2c Maky According to Different Culture Modes. Microorganisms. 2020 Nov 16;8(11):1791. doi: 10.3390/microorganisms8111791. PMID: 33207645; PMCID: PMC7696429.
7: Boratto PVM, Oliveira GP, Machado TB, Andrade ACSP, Baudoin JP, Klose T, Schulz F, Azza S, Decloquement P, Chabrière E, Colson P, Levasseur A, La Scola B, Abrahão JS. Yaravirus: A novel 80-nm virus infecting <i>Acanthamoeba castellanii</i>. Proc Natl Acad Sci U S A. 2020 Jul 14;117(28):16579-16586. doi: 10.1073/pnas.2001637117. Epub 2020 Jun 29. PMID: 32601223; PMCID: PMC7368276.
8: Hasni I, Decloquement P, Demanèche S, Mameri RM, Abbe O, Colson P, La Scola B. Insight into the Lifestyle of Amoeba <i>Willaertia magna</i> during Bioreactor Growth Using Transcriptomics and Proteomics. Microorganisms. 2020 May 21;8(5):771. doi: 10.3390/microorganisms8050771. PMID: 32455615; PMCID: PMC7285305.
9: Rémy B, Plener L, Decloquement P, Armstrong N, Elias M, Daudé D, Chabrière É. Lactonase Specificity Is Key to Quorum Quenching in <i>Pseudomonas aeruginosa</i>. Front Microbiol. 2020 Apr 24;11:762. doi: 10.3389/fmicb.2020.00762. PMID: 32390993; PMCID: PMC7193897.
10: El Karkouri K, Couderc C, Decloquement P, Abeille A, Raoult D. Rapid MALDI- TOF MS identification of commercial truffles. Sci Rep. 2019 Nov 27;9(1):17686. doi: 10.1038/s41598-019-54214-x. PMID: 31776417; PMCID: PMC6881316.
11: Kieu HT, Pham TPT, Lo CI, Alibar S, Bréchard L, Armstrong N, Decloquement P, Diallo A, Sokhna C, Million M, Lagier JC, Raoult D, Tidjani Alou M. Weizmannia faecalis sp. nov., isolated from a human stool sample. Arch Microbiol. 2022 Sep 10;204(10):612. doi: 10.1007/s00203-022-03229-6. PMID: 36087123. 1: Diene SM, Pontarotti P, Azza S, Armstrong N, Pinault L, Chabrière E, Colson P, Rolain JM, Raoult D. Origin, Diversity, and Multiple Roles of Enzymes with Metallo-β-Lactamase Fold from Different Organisms. Cells. 2023 Jun 30;12(13):1752. doi: 10.3390/cells12131752. PMID: 37443786; PMCID: PMC10340364.
12: Azour A, Al-Bayssari C, Pinault L, Azza S, Rolain JM, Diene SM. <i>clbP</i> Gene, a Potential New Member of the β-Lactamase Family. Int J Mol Sci. 2022 Dec 9;23(24):15642. doi: 10.3390/ijms232415642. PMID: 36555283; PMCID: PMC9778894.
13: Maatouk M, Ibrahim A, Pinault L, Armstrong N, Azza S, Rolain JM, Bittar F, Raoult D. New Beta-lactamases in Candidate Phyla Radiation: Owning Pleiotropic Enzymes Is a Smart Paradigm for Microorganisms with a Reduced Genome. Int J Mol Sci. 2022 May 13;23(10):5446. doi: 10.3390/ijms23105446. PMID: 35628255; PMCID: PMC9145738.
14: Diene SM, Pinault L, Baron SA, Azza S, Armstrong N, Hadjadj L, Chabrière E, Rolain JM, Pontarotti P, Raoult D. A metallo-β-lactamase enzyme for internal detoxification of the antibiotic thienamycin. Sci Rep. 2021 May 12;11(1):10062. doi: 10.1038/s41598-021-89600-x. PMID: 33980996; PMCID: PMC8115136.
15: Colson P, Pinault L, Azza S, Armstrong N, Chabriere E, La Scola B, Pontarotti P, Raoult D. A protein of the metallo-hydrolase/oxidoreductase superfamily with both beta-lactamase and ribonuclease activity is linked with translation in giant viruses. Sci Rep. 2020 Dec 10;10(1):21685. doi: 10.1038/s41598-020-78658-8. PMID: 33303919; PMCID: PMC7729979.
16: Diene SM, Pinault L, Armstrong N, Azza S, Keshri V, Khelaifia S, Chabrière E, Caetano-Anolles G, Rolain JM, Pontarotti P, Raoult D. Dual RNase and β-lactamase Activity of a Single Enzyme Encoded in Archaea. Life (Basel). 2020 Nov 14;10(11):280. doi: 10.3390/life10110280. PMID: 33202677; PMCID: PMC7697635.
1: Osman IO, Caputo A, Pinault L, Mege JL, Levasseur A, Devaux CA. Identification and Characterization of an HtrA Sheddase Produced by <i>Coxiella burnetii</i>. Int J Mol Sci. 2023 Jun 30;24(13):10904. doi: 10.3390/ijms241310904. PMID: 37446087; PMCID: PMC10342153.